El equipo es capaz de realizar 40 billones de operaciones por segundo. En Latinoamérica, ocupa el tercer lugar después de Brasil y México.
El Instituto Pasteur de Montevideo y el Centro Nacional de Supercomputación usan la biosimulación para estudiar al virus Sars-Cov2 con el objetivo de compartir sus descubrimientos con la comunidad científica internacional.
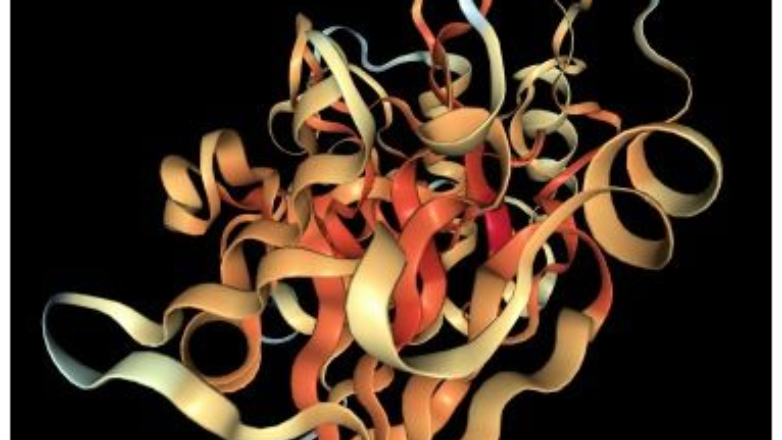
Entre algunos de los trabajos, los centros uruguayos hicieron una simulación para conocer la estructura, dinámica y reproducción de la principal proteína del virus.
Para ello, replicaron su primer contacto con la célula humana.
La infección ocurre cuando la proteína «Spike» (de color naranja), ubicada en la superficie del virus, se acopla a una parte de la célula (de color violeta) para ingresar en ella, introducir su material genético y reproducirse.
Sergio Pantaro, responsable del Laboratorio de Simulaciones Biomoleculares del Instituto Pasteur, explicó que su equipo viene trabajando con estas simulaciones hace ya varios años. La novedad es que contra el coronavirus lograron aumentar su velocidad entre 100 y 1.000 veces.
Al ser una «representación simplificada de las cosas», lograron hacerlas a un costo computacional mucho más bajo porque lograron descartar la información irrelevante y enfocarse en la más importante.
Según Pantaro, eso es posible gracias a la colaboración del Centro Nacional de Supercomputación (Cluster-UY), que «provee servicios de alto desempeño» por medio de su infraestructura informática.
«Son equipos informáticos de gran porte. Proveen la posibilidad de disponer de muchos recursos de cómputo para su uso en simultaneo», cuenta Sergio Nesmachnow, docente grado cinco de la Facultad de Ingeniería de la Universidad de la República. El profesor explicó que mediante el uso coordinado en simultáneo se reduce el tiempo de ejecución y se logra una capacidad de procesamiento de 40 billones de operaciones por segundo.
El Centro Nacional de Supercomputación está construido bajo una arquitectura de cómputo denominada «cluster», que es esa agregación de recursos individuales de procesamiento. Juntos logran 1.120 núcleos de CPU y 100.352 núcleos gráficos GPU. En otros términos, es «10.000 veces más poderoso que una computadora tradicional», comenta el grado cinco.
La biosimulación traduce datos biológicos en ecuaciones matemáticas para crear imágenes digitales de los virus, bacterias y células. En la práctica, esta tecnología ahorra tiempo y dinero, y evita que se haga ensayos en animales.y humanos. La materia prima son coordenadas del átomo, es decir, información sobre dónde están en el espacio. Por medio de cálculos, la máquina predice sus movimientos e interacciones con otros átomos.
«Esperamos que para fines de mayo tengamos los resultados de las simulaciones de todas las proteínas del virus», cuenta Pantaro. Los hallazgos serán compartidos en una base de datos internacional, la cual recopila información sobre estas proteínas para desarrollar avances en la lucha contra el coronavirus. De esa manera, «aportamos un granito para que otros hagan un ladrillo».
Fuente: El Observador